Published at 2020-06-22 09:55
Author:zhixy
View:797
重组是一个基本的生物学过程,例如,可以通过在整个种群中扩散遗传物质来增加病毒或细菌的致病性。重组可导致了镶嵌序列的产生,其中位点之间的进化历史可能不同。 在对一组序列进行系统发育分析时,违反类似树的进化假设(纵向遗传)会导致严重的后果。事实上,由于序列的演化不能用进化树来描述,这可能导致高估或低估分支长度等问题。 因此,对于给定的一组对齐序列来说,一个重要的问题是确定是否可能发生重组。
检验统计量——成对同质指数(pairwise homoplasy index, 𝚽w),是在各种各样的情况下区分反复突变和重组的最佳方法之一。
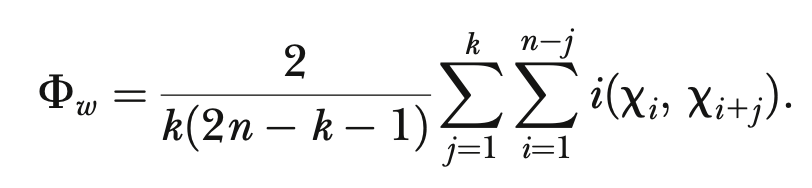
Phi检验是一种简单、快速和统计上有效的重组检验。 它的性能与基于聚结的方法(如LDHat)相当,但它可以应用于与数百个序列的较大序列比对。
原文献中的程序下载地址已不可用,但作者之一的David Bryant在其实验室的网站上提供了下载。
下载源码编译后,即可使用Phi进行重组检验。
(base) [user@server ~]# Phi
Input file not specified!!! (use -f|-r|-s)
Usage: Phi [-f|-s|-r] Filename [-t] AlignmentType
[-p [#]] [-o] [-v] [-g [#]] [-b [#]]
Options:
-f: Filename = FASTA format
-s: Filename = Strict phylip file
-r: Filename = Relaxed phylip file
-t: AlignmentType = D|A|O where D=DNA
A=AA and O=OTHER [default DNA]
-p: [#] = PHI permutation test [default = FALSE, #=1000]
-w: # = Change default window size [default w = 100]
-o: Report other statistics (NSS and Max Chi^2) [default = FALSE]
-v: Verbose [default = FALSE]
-g: [i] = Print color (scaled) incompatibility matrix (graph.ppm)
i - Image only (no ticks...) [default = FALSE]
(base) [user@server ~]# Phi -f test.fasta
Reading sequence file test.fasta
Found 70 sequences of length 1542
Alignment looks like a valid DNA alignment.
Estimated diversity is (pairwise deletion - ignoring missing/ambig): 21.7%
Found 806 informative sites.
Writing alignment of informative sites to: Phi.inf.sites
Writing list of informative sites to: Phi.inf.list
Calculating all pairwise incompatibilities...
Done: 100.0%
Using a window size of 100 with k as 52
Calculating analytical mean and variance
**p-Value(s)**
----------
PHI (Normal): 3.75e-03
(base) [user@server ~]$
P < 0.05,即测试对齐序列中存在重组现象。
Trevor C. Bruen, Hervé Philippe and David Bryant. A Simple and Robust Statistical Test for Detecting the Presence of Recombination. GENETICS April 1, 2006 vol. 172 no. 4 2665-2681. (DOI:10.1534/genetics.105.048975)
